多組非常態分佈資料之差異檢定與事後比較:R的Kruskal–Wallis檢定與Welch's anova / Non-Parametric Tests for Comparing Many Non-normal Distribution Groups: Kruskal–Wallis test and Welch's anova in R
很多時候我們研究的資料不一定符合常態分佈,例如車禍數量的趨勢符合泊松分佈(Poisson distribution)、產品的生命週期符合韋伯分佈(Weibull distribution),甚至是健康、教育和社會科學中最常見的出現次數(frequency of appearance)這種研究資料都不是常態分佈(請見Bono等人在2017年的回顧文章)。
當要比較的多組資料為常態分佈時,我們可以用單因子變異數(one-way ANOVA)。但若多組資料並非常態分佈時,我們則要使用中位數的ANOVA:Kruskal–Wallis檢定以及事後比較Dunn檢定。而若各組資料之間變異數不同質時,則要用Welch's anova跟事後比較Games-Howell檢定。
為了方便使用,我將多組非常態分佈資料檢定的方法以R撰寫成一個腳本Non-Parametric Tests for Comparing Many Non-normal Distribution Groups.R供大家使用。以下將介紹如何使用這個腳本來進行多組非常態分佈的差異檢定。
非常態分佈的資料 / Non-normal distribution data
在健康、教育和社會科學等領域的研究中所蒐集的資料,很多時候都是非常態分佈。Non-Normal Distributions in the Real World介紹了非常態分佈的常見資料,像是人類的行為模式,例如收到發貨單跟付款之間的差距天數,或是受到物理法則影響的資料,像是鍍鋅厚度的分佈。
How do I determine whether my data are normal?這篇講述了四種判斷資料是否為常態分佈的方法,摘要如下:
- 觀察疊加了常態分佈曲線(normal curve)的直方圖(histogram),看資料是否非常偏離常態分佈曲線。
- 觀察偏態值(Skewness)是否遠離0。0表示為常態分佈。
- 使用常態分佈檢定Kolmogorov-Smirnov test (K-S)與Shapiro-Wilk (S-W)檢定。
- 使用「常態分位圖」(Normal Q-Q Plot),觀看資料是否落於常態分佈線上。
有時候資料可能會是常態分佈,但因為某些問題導致它看起來是非常態分佈。Statistics How To的Non Normal Distribution歸納了四個可能會讓資料呈現非常態分佈的理由:
- 資料包含異常值(outliers):如果資料包含了異常值,則資料分佈會變成偏態(skewed)。有母數統計的核心平均數(mean)特別容易收到異常值的影響。移除異常值之後再來分析看看。
- 資料分佈是由多組常態分佈的資料組成:這會造成雙峰(bimodal)或多峰(multimodal)的分佈。例如,下圖包含了兩組常態分佈的測試結果,這讓資料呈現了雙峰分佈。
(圖片來源:Statistics How To) - 資料不足:如果資料數量不夠大,則會讓常態分佈的資料看起來完全分散。舉例來說,課堂測驗結果一般是常態分佈。如果我們用極端的例子來看,只隨機抽取三位學生並將之繪製統計圖表,則結果並不會是常態分佈。可能會是得到均勻分佈(uniform distribution),如62、62、63;也可能會得到偏態分佈,例如80、92、99。蒐集更多資料再來分析看看吧。
- 錯誤的統計圖表誤導。通常是X軸或Y軸的單位、區間設定錯誤,導致直方圖出現偏態。請確認直方圖正確的繪製再來分析資料吧。
若你的資料確實是非常態分佈,那麼你可能需要用這篇的方法來分析這些資料。
R腳本與執行環境 / R script and environment
R腳本必須要在有R的環境下執行。我個人使用的是在Windows上執行的RStudio,版本是1.1.383。在安裝RStudio的時候會一同安裝R環境。我使用的R的版本是3.4,此外還會用到多個套件,套件的安裝全部寫在R的腳本裡。
- R腳本:Non-Parametric Tests for Comparing Many Non-normal Distribution Groups.R
- RStudio:下載RStudio Desktop網頁
此腳本主要改編自An R Companion for the Handbook of Biological Statistics的Kruskal–Wallis Test,後面加上的Welch's anova則是來自於同網站的One-way Anova,事後多重比較Games-Howell檢定則是來自於RPubs。
因為R的版本更新的非常頻繁,你閱讀這篇的時候安裝的不一定是跟我一樣的R 3.4版,可能會有版本差異的問題。若安裝套件時發生了版本不符合的問題,請看Ubuntu中舊版R安裝套件的方法。關於R的版本問題,請看我之前學習R的感想。
參考資料 / Reference
此腳本主要改編自An R Companion for the Handbook of Biological Statistics的Kruskal–Wallis Test,後面加上的Welch's anova則是來自於同網站的One-way Anova,事後多重比較Games-Howell檢定則是來自於RPubs。
至於Kruskal–Wallis檢定的理論基礎請見John H. McDonald所著的Kruskal–Wallis test,裡面介紹了Kruskal–Wallis檢定的適用時機以及與它與單因子變異數的差別,該篇也建議Kruskal–Wallis檢定應該只用在排序資料上,像是社會科學中權力階層的上下位關係或是發展階段先後順序。Welch's anova的理論基礎請見Jim Frost所著的Benefits of Welch’s ANOVA Compared to the Classic One-Way ANOVA。大部分時候Welch's anova都是被視為當ANOVA違反變異數同質性檢定時的替代方案,但它也可以在Kruskal–Wallis檢定違反變異數同質性檢定的時候使用。
實作:多組非常態分佈同質性資料之中位數差異檢定 / Practice: Test for difference in median among many homoscedastic groups
| Group 1 | Group 2 | Group 3 |
|---|---|---|
| 1 | 10 | 19 |
| 2 | 11 | 20 |
| 3 | 12 | 21 |
| 4 | 13 | 22 |
| 5 | 14 | 23 |
| 6 | 15 | 24 |
| 7 | 16 | 25 |
| 8 | 17 | 26 |
| 9 | 18 | 27 |
| 46 | 37 | 28 |
| 47 | 58 | 65 |
| 48 | 59 | 66 |
| 49 | 60 | 67 |
| 50 | 61 | 68 |
| 51 | 62 | 69 |
| 52 | 63 | 70 |
| 53 | 64 | 71 |
| 342 | 193 | 72 |
(表格來源:Handbook of Biological Statistics)
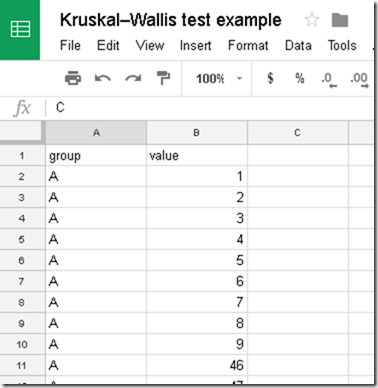
以下我用John H. McDonald在介紹Kruskal–Wallis檢定的時候使用的資料作為例子,以此來示範如何使用R腳本Non-Parametric Tests for Comparing Many Non-normal Distribution Groups.R來進行Kruskal–Wallis檢定。
資料格式 / Data format
R腳本需要輸入的資料必須是CSV格式,以逗點(,)隔開欄位,而第一行必須是group跟 value ,group欄位擺放組別名稱,value欄位則是擺放數字資料。請務必依照這個格式來建立資料,這樣才能用在R腳本中。
請下載CSV格式,待會兒就來使用。
執行R腳本 / Excute R script
請在Non-Parametric Tests for Comparing Many Non-normal Distribution Groups.R檔案按右鍵,在右鍵選單中的「開啟檔案」點選RStudio。
接著RStudio會開啟並載入R腳本。我們在腳本的位置按熱鍵Ctrl + A全選腳本文字,然後按下上方的「Run」按鈕。
選擇剛剛下載的CSV檔案,然後按下「開啟」。
如果是第一次執行,R環境需要安裝大量的套件,大概會花個10分鐘左右,請耐心等候。第二次執行的時候,大概30秒內可以完成。最後看到左下角的Console有出現「Finish」就表示執行完成。
在CSV檔案的目錄底下會出現以CSV檔案為名、並加上日期時間與輸出資料後綴的統計結果檔案,包括:
- 直方圖(histogram):檔案名稱例子為Kruskal–Wallis test example - data_0127-2042_ histogram.png
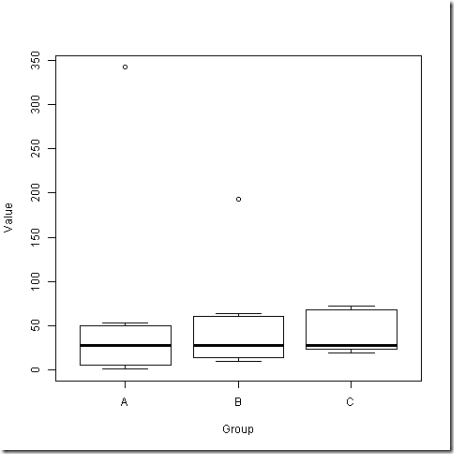
- 箱型圖(boxplot):檔案名稱例子為Kruskal–Wallis test example - data_0127-2042_ boxplot.png
- 檢定結果:Kruskal–Wallis test example - data_0127-2042_ result.txt
以下我們以Kruskal–Wallis檢定結果檔案為主,其他兩個統計圖表為輔,來解說Kruskal–Wallis檢定結果吧。
檢定結果 / Result
在result.txt為後綴的檔案中記錄著分析的過程,以下我就各個段落進行解說:
檔案名稱 / File
### File:
C:\data\Kruskal–Wallis test example - data.csv記錄該結果是來自於那個檔案。
敘述統計 / Descriptive statistics
### Medians and descriptive statistics
group n mean sd min Q1 median Q3 max
1 A 18 43.5 77.77513 1 5.25 27.5 49.75 342
2 B 18 43.5 43.69446 10 14.25 27.5 60.75 193
3 C 18 43.5 23.16755 19 23.25 27.5 67.75 72敘述統計會列出各個組別的組別名稱(group)、個數(n)、平均數(mean)、標準差(sd)、最小值(min)、下四分位數(Q1)、中位數(median)、上四分位數(Q3)、最大值(max)。就像我在資料的中心與離度所強調的,在說明敘述統計的時候,最好搭配圖表一起看。
histogram.png是各組的直方圖,注意Y軸為所佔比例,而非資料的個數。從這邊可以看到A組跟B組各有一個異常值,導致這兩組的資料明顯呈現右偏。
boxplot.png是各組的箱型圖,我們可以看到三組的五數綜合比較。儘管三組的中位數都相同,但A組跟B組的離群值讓三組的資料分佈有所差異。關於箱型圖的閱讀方法,請看用R畫箱型圖。
常態性檢定 / Normality test
### Normality test
Group: A, Shapiro-Wilk normality test W = 0.4856823 p-value = 6.035973e-07
Group: B, Shapiro-Wilk normality test W = 0.6784267 p-value = 4.530191e-05
Group: C, Shapiro-Wilk normality test W = 0.7404266 p-value = 0.0002453774這裡測試各組的資料分佈是否為常態分佈。根據柴惠敏的建議,若該組的樣本個數小於50,則是使用Shapiro-Wilk常態檢定,檢定統計量為W值;若該組的樣本數在50以上,則是使用Kolmogorov-Smirnov (K-S)常態檢定,檢定統計量為D值。STHDA推薦使用Shapiro-Wilk檢定,因為它的統計檢定力(power)高於Kolmogorov-Smirnov檢定。這邊R腳本會依照該組的樣本個數來自動判斷要使用的檢定法。
這兩個檢定的虛無假設都是該組資料為常態分佈。因此若p值在0.05以下,則表示該組資料並非常態分佈。以這個例子來說,A組的p值為6.035973e-07,有e表示是科學記號格式,意思是6.035973乘以-10的7次方,也就是非常非常小的小數,低於顯著水準0.05,因此A組資料並非常態分佈。另外C組的p值為0.0002453774,低於顯著水準0.05,因此也不是常態分佈。
一般來說,無母數檢定並不會特別去檢定資料分佈是否非常態。這個常態性檢定僅供參考即可。
變異數同質性檢定 / Test for Homogeneity of Variance
### Test for Homogeneity of Variance
Levene's Test for Homogeneity of Variance (center = median)
Df F value Pr(>F)
group 2 0.6864 0.508
51
Data are homoscedastic. Excute Kruskal–Wallis test.這邊我們使用Levene變異數同質性檢定中無母數檢定的方法來檢定三組的變異數是否相同。請看「Pr(>F)」這一欄的顯著性是否在0.05以上。若在0.05以上使用的是Kruskal–Wallis檢定,否則使用Welch's anova。
在本例中,因為三組資料變異數同質,因此進行Kruskal–Wallis檢定。
Kruskal–Wallis檢定 / Kruskal–Wallis test
### Kruskal–Wallis test for equal variances
Kruskal-Wallis rank sum test
data: value by group
Kruskal-Wallis chi-squared = 7.3553, df = 2, p-value =
0.02528
Eta squared: 0.138779831861593檢定結果的卡方統計檢定量請看Kruskal-Wallis chi-squared,為7.3553。自由度df為2。各組之間是否有顯著差異則要看p-value,若在顯著水準0.05以上,則表示各組之間的中位數沒有顯著差異,否則表示各組之間的中位數有顯著差異。本例的p-value為0.02528,表示各組之間的中位數達到顯著差異。
最後的Eta squared是表示分組變項group對於value的影響力,也就是關聯強度。計算方式來自於how2stats的Kruskal-Wallis - SPSS教學。關於Eta squared的說明請看我介紹ANOVA的這篇。Eta squared的判斷準則如下:
- 0.059 > eta squared >= 0.01 低度關聯強度
- 0.138 > eta squared >= 0.059 中度關聯強度
- eta squared >= 0.138 高度關聯強度
以本例的0.138779831861593為例,是屬於高度關聯強度。
Dunn事後多重比較檢定 / Dunn Post Hoc test
### Dunn test
Comparison Z P.unadj P.adj
1 A - B -1.356036 0.17508782 0.17508782
2 A - C -2.712071 0.00668642 0.02005926
3 B - C -1.356036 0.17508782 0.26263172若Kruskal–Wallis檢定有顯著差異,則繼續查看Dunn檢定的結果。Dunn檢定將各組之間兩兩比較,以此查看各組之間是否有顯著差異。本例中有三個組別「A」、「B」、「C」,因此共有「A - B」、「A - C」跟「B - C」三種組合可供比較。其中第二組比較「A - C」結果中的調整後P值(P.adj) 0.02小於0.05,而Z值為-2.712071小於0,表示後者組別「C」顯著大於前者組別「A」。而其他兩組比較則沒有顯著差異。
這樣子就完成了Kruskal–Wallis檢定跟Dunn事後多重比較囉。
實作:多組非常態分佈異質性資料之中位數差異檢定 / Practice: Test for difference in mean among many heteroscedastic groups
許多使用Kruskal–Wallis檢定的研究者常常忘記Kruskal–Wallis檢定有各組資料為同質性的假設。當各組的個數相同,或是分佈類似時,各組資料常常具有同質性(homoscedastic)。但若各組的樣本數差異過大時,很容易就會發生各組資料為異質性(heteroscedastic)的情況,這時候就要改用Welch's anova來分析。我寫的R腳本中會以無母數的Levene檢定來做各組資料是否為同質性的檢定,以此來判斷要選擇用Kruskal–Wallis檢定還是Welch's anova。以下我們就來看看這種情況會怎麼分析。
異質性的資料 / Heteroscedastic data
這份資料是來自於Real Statistics Using Excel的Welch's ANOVA Test的例子。我們的格式也一樣要有group跟value,然後請下載CSV格式做準備吧。
待會會使用這個CSV檔案來分析。
執行R腳本 / Excute R script
操作方法跟上述相同,在此我們就不贅述。若順利分析完成,最後一樣可以獲得三個檔案:
- 直方圖:Welchs ANOVA Test example - data_0128-1514_ histogram.png
- 箱型圖:Welchs ANOVA Test example - data_0128-1514_boxplot.png
- 檢定結果:Welchs ANOVA Test example - data_0128-1514_ result.txt
以下我們依然是來從檢定結果檔案來說明分析的結果吧。
檢定結果 / Result
在result.txt為後綴的檔案中記錄著分析的過程,以下我就各個段落進行解說:
檔案名稱 / File
### File
C:\data\Welchs ANOVA Test - data.csv這是本次分析的檔案。
敘述統計 / Descriptive statistics
### Medians and descriptive statistics
group n mean sd min Q1 median Q3 max
1 New 10 43.00000 4.027682 38 40.25 42 44.75 50
2 Old 9 33.44444 9.302031 22 27.00 31 40.00 50
3 Control 8 35.75000 16.298554 16 22.75 34 46.75 60敘述統計中可以看到這次的資料有三組「New」、「Old」跟「Control」,三組的個數(n)、平均數(mean)、標準差(sd)都不一樣。
在以histogram.png後綴的直方圖裡面可以看到三組的分佈。Control的分佈較廣,Old的資料偏小且分散,而New的資料則較大且集中。
在以boxplot.png後綴的箱型圖裡面可以清楚比較三組的分佈。可以看到New大部分都大於Old,而Control則是涵蓋前兩組。
常態性檢定 / Normality test
### Normality test
Group: New, Shapiro-Wilk normality test W = 0.9239325 p-value = 0.3909235
Group: Old, Shapiro-Wilk normality test W = 0.9327685 p-value = 0.5081456
Group: Control, Shapiro-Wilk normality test W = 0.9419088 p-value = 0.6299536常態性檢定中,因為各組資料個數都小於50,因此使用的是Shapiro-Wilk檢定。三組的p值都大於顯著水準0.05,表示三組皆為常態分佈。因為這個例子是用來跟有母數統計ANOVA做對比用,因此資料本身是為常態分佈。
變異數同質性檢定 / Test for Homogeneity of Variance
### Test for Homogeneity of Variance
Levene's Test for Homogeneity of Variance (center = median)
Df F value Pr(>F)
group 2 6.5194 0.005478 **
24
---
Signif. codes: 0 ‘***’ 0.001 ‘**’ 0.01 ‘*’ 0.05 ‘.’ 0.1 ‘ ’ 1
Data are heteroscedastic. Excute Welch's anova.在Levene檢定中,Pr(>F)的數值0.005478小於顯著水準0.05,表示各組資料為異質性,因此接下來將使用Welch's anova進行分析。
Welch's anova
### Welch’s anova for unequal variances
One-way analysis of means (not assuming equal variances)
data: value and group
F = 4.3153, num df = 2.0, denom df = 11.7, p-value = 0.03947在Welch's anova中檢定的是各組的平均數是否相同,檢定統計量F值為4.3153,而顯著性p值(p-value)為0.03947,小於顯著水準0.05,表示三組資料之間有顯著差異。
Games-Howell事後多重比較 / Games-Howell Post-Hoc Test
### Games-Howell Post-Hoc Test
### Oneway Anova for y=value and x=group (groups: New, Old, Control)
Omega squared: 95% CI = [NA; .36], point estimate = .08
Eta Squared: 95% CI = [0; .32], point estimate = .15
SS Df MS F p
Between groups (error + effect) 474.28 2 237.14 2.11 .143
Within groups (error only) 2697.72 24 112.41
### Post hoc test: games-howell
diff ci.lo ci.hi t df p
Old-New -9.56 -18.65 -0.46 2.85 10.66 .040
Control-New -7.25 -24.26 9.76 1.23 7.69 .472
Control-Old 2.31 -15.41 20.02 0.35 10.84 .934Eta Squared的點估計值為0.15,表示高度關聯強度。然後我們看「### Post hoc test: games-howell」以下的部分,可以看到「Old」、「New」、「Control」三組的比較結果。
在「Old-New」的比較中,p值(p)為0.04,小於顯著水準0.05,而diff為-9.56,表示前者「Old」比後者「New」還小。另一方面,「Control-New」跟「Control-Old」的比較中,p值都超過0.05,表示兩組比較沒有顯著差異。
這樣子就完成了Welch's anova跟Games-Howell事後多重比較囉。
結語 / Conclusion
本篇以R腳本實作了Kruskal–Wallis檢定跟Dunn事後多重比較以及Welch's anova跟Games-Howell事後多重比較這兩套無母數統計方法,可以用來分析非常態分佈的多組資料之間是否有顯著差異。這個方法整理完之後,應該可以對社會科學、行為研究帶來很大的幫助。
我本來是想要用SPSS來完成這個分析,但看了最完整的how2stats的Kruskal-Wallis - SPSS教學後,發現它不僅操作起來非常複雜,而且也只實作了最基本的Kruskal–Wallis檢定,更別說變異數同質性檢定、常態分佈檢定、直方圖跟箱型圖的繪製等等。後來我看了幾篇用R實作的教學,發現R做起來不僅容易許多,而且輸出資料也很容易客製化,甚至要用Kruskal–Wallis檢定還是Welch's anova也都可以用程式判斷,所以這次我就把它整理成更好用的R腳本,提供給大家使用。這次的R腳本比上次在做循序樣式探勘的時候進步許多,蠻接近我理想中的R的用法。
這篇在撰寫的時候,本來只有Kruskal–Wallis檢定跟Dunn事後多重比較,但很多文獻提到了非同質性時的處理方法,又有在講資料是否為常態分佈的檢定,我就一邊看一邊加入各種功能,最後花了兩天的時間才整理完這份R腳本以及這篇教學,花的時間意外的多了很多。
之後有時間的話我也想依照這份腳本做成一份ANOVA分析用的R腳本,同時也將資料違反同質性假設(是異質性)的時候改用Welch's anova的方法也納入考量,這樣應該會讓ANOVA好用許多。
這篇多組非常態分佈資料的差異檢定的整理就到這裡為止。我本身並非統計專業,只是因為分析資料時會用到,所以看文獻來將它整理一下。如果有什麼統計上的建議,歡迎在下面留言提出指教。當你分析的資料也是非常態分佈的時候,那這篇應該會對你有些幫助。如果你覺得我整理的不錯的話,請幫我在AddThis分享工具按讚、將這篇分享到Facebook等社群媒體吧!感謝你的耐心閱讀,讓我們下一篇見。














修正了使用RScript Caller呼叫時的問題
回覆刪除現在它也可以正常使用RScript Caller囉
http://blog.pulipuli.info/2018/02/rrscript-caller-runing-r-script.html
中間關於eta squared的 中度關聯的地方 0.59應該是0.059對吧?
回覆刪除已經修正,謝謝您的提醒。
刪除布丁大大您好~
回覆刪除想請問一下那如果同時違反常態性假設跟同質變異數假設有什麼檢定以及事後檢定可以用嗎?
非常好奇~因為現在碰到的實務資料就是這樣,但有想以科學的方式去解釋他們~"~
你好,
刪除這篇就是非常態分佈的差異檢定法,你要不要先用這篇的做法做做看?
你要不要再看一看這篇的做法,然後針對這篇做法中不懂的地方再提問吧?
嘿~布丁大大
回覆刪除不好意思,可能是我沒講清楚或是是我認知錯誤還請您幫我看一下
符合常態且變異數同質>ANOVA
符合常態且變異數異質>Welch's ANOVA
不符合常態且變異數同質>K-W test
不符合常態且變異數異質>???
我是想問最後一個如果兩者都不相符的話該採用什麼檢定
您好,
刪除我覺得應該重新檢討整個研究跟資料蒐集方法,甚至是你分析的方式。
好的~非常感謝您!
刪除加油!
刪除想問布丁
回覆刪除Games-Howell Post-Hoc Test 的套件是用哪一個的
我用的是userfriendlyscience 裡面的
但結果後面沒有
Old-New
Control-New
Control-Old
你好,
刪除你是用同一份資料,然後缺少多重比較的結果嗎?
"0.138 > eta squared >= 0.59 中度關聯強度" 這邊應該改成 "0.138 > eta squared >= 0.059 中度關聯強度"
回覆刪除您好,
刪除感謝您的指正,已經修正了。
您好,感謝你提供如此方便的腳本!
回覆刪除想請在腳本中後最後使用了 Welch's anova,p值也小於0.05但卻沒有事後統計,可能是甚麼問題呢?
啾啾您好,
刪除1. 請問顯示的訊息是什麼?
2. group的水準是三個嗎?
Error in library(userfriendlyscience) :
刪除there is no package called ‘userfriendlyscience’
使否是因為我沒安裝userfriendlyscience這個packages
抱歉,真的是我耍笨沒裝userfriendlyscience
回覆刪除再次感謝你~
To 啾啾,
刪除userfriendlyscience套件已經被移除了,我們沒辦法用`install.packages("userfriendlyscience")`安裝它
https://cran.r-project.org/web/packages/userfriendlyscience/index.html
替代做法是從GitHub安裝
目前userfriendlyscience套件有典藏在GitHub https://github.com/Matherion/userfriendlyscience
如果要從GitHub安裝R套件,我們需要安裝githubinstall套件
以下是我在ubuntu-20.04-desktop-amd64裡面的安裝步驟:
1. 先在系統的terminal裡面安裝Ubuntu必要套件
sudo apt-get install libcurl4-openssl-dev libssl-dev libgsl0-dev libfontconfig1-dev -y
2. 進入R環境
$ R
3. 安裝githubinstall套件
> install.packages("githubinstall")
4. 從GitHub安裝userfriendlyscience套件
> githubinstall("Matherion/userfriendlyscience")
這一步我裝了一個小時
5. 確認userfriendlyscience套件安裝成功
> library(userfriendlyscience)
到此為主算是完成。
----
步驟繁瑣,請根據系統回報的訊息一一確認。
加油。
今天遇到一個特別的情況,是Windows 10無法安裝套件
回覆刪除如果遇到無法安裝套件的情況,請用系統管理員身份開啟RStudio,應該就可以安裝普通的套件了。
您好,想請問在Kruskal-wallis test要改變實驗材料中什麼樣的條件,才能讓原本跑出來結果沒有顯著差異變成有顯著差異(p<0.05)呢?謝謝您
回覆刪除想要在專題實務安全下庄 您好,
刪除如果想要讓多組的中位數之間差異大到出現顯著差異,有幾個方法可以嘗試:
1. 試著讓某一組的分數中位數更高,或讓某一組的分數中位數更低,加大組跟組之間的差異。
2. 增加各組內的數量
3. 減少組的數量
試試看
非常感謝您 有您真好
刪除想要在專題實務安全下庄 您好,
刪除要達到統計上的顯著,意思是排除掉隨機發生的可能性,其實本來就不是一件容易的事情。
如果你讓某件事情放著自然發生,一般情況下很難達到顯著水準。
反之,如果出現顯著,表示這件事情的確有點「異常」。
加油。
您好,如果我在Kruskal Wallis test跑出來的統計數據為0.072,感覺離顯著水準0.05蠻相近!那想藉此提問,Kruskal Wallis test 是否有個正負的標準誤的範圍呢?謝謝您
回覆刪除To 想要在專題實務安全下庄,
刪除alpha level顯著水準是一個門檻,表示你拒絕虛無假設(各組相同)的機率。
一般來說,學界的顯著水準設為0.05,表示只接受p值在0.05以下。
至於0.072行不行嘛...
你去跟你老師討論看看吧...
好的,謝謝您!
刪除To 想要在專題實務安全下庄,
刪除加油!